Über uns
Die Erhebung von Genomdaten von Patient:innen ist eine Schlüsseltechnologie in der biomedizinischen Forschung. Diese Art von Daten werden zunehmend auch in der Diagnostik und im Rahmen von personalisierten Therapien erzeugt. So entstehen aktuell an einer wachsenden Anzahl von Forschungsstandorten in Deutschland umfangreich charakterisierte Datensätze. Für eine optimale Nutzung dieser Daten in der Forschung ist eine weitreichende Integration dieser lokalen Datensätze unerlässlich. Nur so können moderne Computermethoden wie maschinelles Lernen oder künstliche Intelligenz ihr volles Potential entfalten. Dabei muss das Bedürfnis, Daten offen und FAIR für die Forschungsgemeinde zu erschliessen immer mit dem Schutz der Privatsphäre der Patient:innen abgewogen werden.
Um diesen Balanceakt zu ermöglichen, baut GHGA (German Human Genome-Phenome Archive) eine nationale Infrastruktur für humane Genomdaten auf.
Diese soll es erlauben, hochsensible Genomdaten in einem einheitlichen, datenschutzkonformen Rahmen zusammenzuführen, zu speichern und zu analysieren.
Als nationaler Knoten des föderierten Europäischen Genom-Phänom-Archivs (EGA) kann GHGA spezifische nationale Regelungen zum Datenschutz folgen und gleichzeitig eng an internationale Dateninfrastrukturen angebunden sein. Dies macht die Datensätze leicht auffindbar und optimal für die nationale und internationale Forschung nutzbar.
GHGA wird auch die Wünsche der Forschungsgemeinde nach effizienten, benutzerfreundlichen Analysen im großen Maßstab berücksichtigen. Die Validierung von Analyseergebnisse auf anderen Kohorten ist ein weitere Schlüsselressource, die GHGA anbieten wird. GHGA setzt dabei auf existierenden nationalen Omics-Datenlieferanten und deren IT-Infrastrukturen auf, um eine harmonisierte, interoperable Infrastruktur zu schaffen.
Ziele
- Bereitstellung eines nationalen Langzeitarchivs humaner Omics-Daten
- Adressierung rechtlicher und ethischer Hindernisse für Datenaustausch in der Forschung
- Erhöhung der FAIRness von Omics-Daten (Einbettung in nationale und internationale Dateninfrastrukturen)
- Entwicklung und Bereitstellung maßgeschneiderter Datenportale, um spezifischen Anforderungen an Datensätzen und Analysetools gerecht zu werden
- Sensibilisierung für die effiziente und verantwortungsvollen Nutzung und Verwaltung von Omics-Daten
- Demokratisierung des Zugriffs und der Analyse umfangreicher Omics-Daten über eine Cloud-basierte Analyseplattform
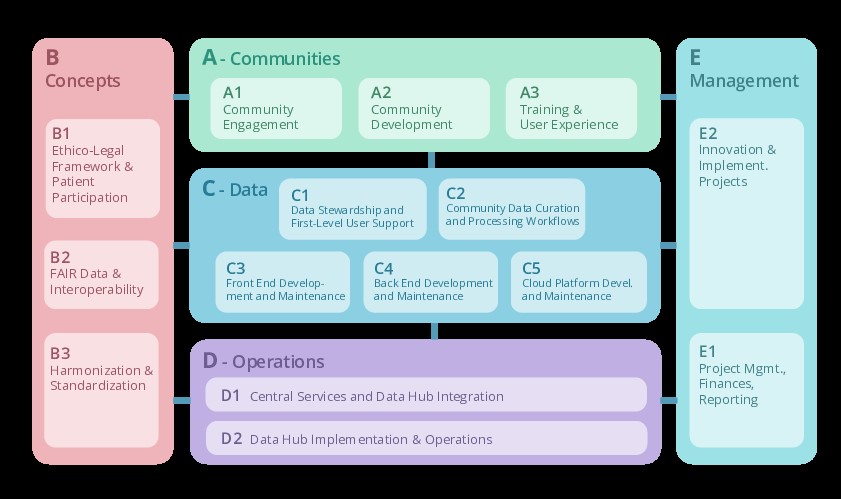
Aufgabenbereich

Prof. Dr. Oliver Stegle
Sprecher des Konsortiums

Deutsches Krebsforschungszentrum
Antragsstellende Institution

Oliver Kohlbacher
Board of Directors

Jan Korbel
Board of Directors

Eva Winkler
Board of Directors
(Mit-)Antragstellende Institutionen und (Co-)Sprecher:innen:
- Peer Bork – EMBL, Heidelberg
- Ivo Buchhalter – DKFZ, Heidelberg
- Andreas Dahl – TU Dresden, Dresden
- Julien Gagneur – TUM, München
- Wolfgang Huber – EMBL, Heidelberg
- Daniel Hübschmann – DKFZ, Heidelberg
- Oliver Kohlbacher – EKUT, Tübingen
- Jan Korbel – EMBL, Heidelberg
- Martin Lablans – DKFZ, Heidelberg
- Peter Lichter – DKFZ, Heidelberg
- Fruzsina Molnár-Gábor – HAdW, Heidelberg
- Susanne Motameny – UzK, Köln
- Sven Nahnsen – EKUT, Tübingen
- Uwe Ohler – MDC, Berlin
- Stephan Ossowski – UKT, Tübingen
- Annette Peters – HMGU, München
- Olaf Rieß – UKT, Tübingen
- Philip Rosenstiel – UKI, Kiel
- Thorsten Schlomm – CHARITE, Berlin
- Joachim Schultze – DZNE, Bonn
- Oliver Stegle – DKFZ/EMBL, Heidelberg
- Jörn Walter – UdS, Saarbrücken
- Thomas Walter – EKUT, Tübingen
- Stefan Wesner – UzK, Köln
- Juliane Winkelmann – HMGU & TUM, München
- Eva Winkler – UHH, Heidelberg
- Eberhard-Karls-Universität Tübingen (EKUT)
- University Hospital Tübingen (UKT)
- Charité – Universitätsmedizin Berlin (Charité)
- Technische Universität München (TUM)
- Europäisches Laboratorium für Molekularbiologie (EMBL)
- Max Delbrück Center for Molecular Medicine (MDC) Technische Universität
- Dresden (TU Dresden)
- University Hospital Heidelberg (UHH)
- Heidelberger Akademie der Wissenschaften (HAdW)
- Universität zu Köln (UzK)
- Universitätsklinikum Schleswig-Holstein (UKI)
- Helmholtz Zentrum München (HMGU)
- Dt. Zentrum für Neurodeg. Erkrankungen e.V. (DZNE)
- Universität des Saarlandes (UdS)
- German National Cohort (GNC)
Beteiligte Institutionen
- Viktor Achter – UzK
- Dieter Beule – MDC, Berlin
- Benedikt Brors – DKFZ, Heidelberg
- Holm Graessner – UKT, Tübingen
- Michael Hummel – Charité, Berlin
- Dirk Jäger – UHH, Heidelberg
- Jens Krüger – EKUT, Tübingen
- Nisar Malek – UKT, Tübingen
- Thomas Meitinger – HMGU & TUM, München
- Wolfgang E. Nagel – TU Dresden, Dresden
- Julio Saez-Rodriguez – UHH, Heidelberg
- Christoph Schickhardt – UHH, Heidelberg
- Thomas Keane – EMBL-EBI Cambridge, UK
- Mario Fritz & Ninja Marnau – CISPA Saarbrücken
- Stephan Hachinger – LRZ München
- Alice McHardy – HZI Braunschweig
- Stefan Fröhling & Hanno Glimm – National Center for Tumor Diseases (NCT), Heidelberg and Dresden

Oliver Stegle
Sprecher des Konsortiums

Deutsches Krebsforschungszentrum
Antragsstellende Institution

Oliver Kohlbacher
Board of Directors

Jan Korbel
Board of Directors

Eva Winkler
Board of Directors
(Mit-)Antragstellende Institutionen und (Co-)Sprecher:innen:
- Peer Bork – EMBL, Heidelberg
- Ivo Buchhalter – DKFZ, Heidelberg
- Andreas Dahl – TU Dresden, Dresden
- Julien Gagneur – TUM, München
- Wolfgang Huber – EMBL, Heidelberg
- Daniel Hübschmann – DKFZ, Heidelberg
- Oliver Kohlbacher – EKUT, Tübingen
- Jan Korbel – EMBL, Heidelberg
- Martin Lablans – DKFZ, Heidelberg
- Peter Lichter – DKFZ, Heidelberg
- Fruzsina Molnár-Gábor – HAdW, Heidelberg
- Susanne Motameny – UzK, Köln
- Sven Nahnsen – EKUT, Tübingen
- Uwe Ohler – MDC, Berlin
- Stephan Ossowski – UKT, Tübingen
- Annette Peters – HMGU, München
- Olaf Rieß – UKT, Tübingen
- Philip Rosenstiel – UKI, Kiel
- Thorsten Schlomm – CHARITE, Berlin
- Joachim Schultze – DZNE, Bonn
- Oliver Stegle – DKFZ/EMBL, Heidelberg
- Jörn Walter – UdS, Saarbrücken
- Thomas Walter – EKUT, Tübingen
- Stefan Wesner – UzK, Köln
- Juliane Winkelmann – HMGU & TUM, München
- Eva Winkler – UHH, Heidelberg
- Eberhard-Karls-Universität Tübingen (EKUT)
- University Hospital Tübingen (UKT)
- Charité – Universitätsmedizin Berlin (Charité)
- Technische Universität München (TUM)
- Europäisches Laboratorium für Molekularbiologie (EMBL)
- Max Delbrück Center for Molecular Medicine (MDC) Technische Universität
- Dresden (TU Dresden)
- University Hospital Heidelberg (UHH)
- Heidelberger Akademie der Wissenschaften (HAdW)
- Universität zu Köln (UzK)
- Universitätsklinikum Schleswig-Holstein (UKI)
- Helmholtz Zentrum München (HMGU)
- Dt. Zentrum für Neurodeg. Erkrankungen e.V. (DZNE)
- Universität des Saarlandes (UdS)
- German National Cohort (GNC)
Beteiligte Institutionen:
- Viktor Achter – UzK
- Dieter Beule – MDC, Berlin
- Benedikt Brors – DKFZ, Heidelberg
- Holm Graessner – UKT, Tübingen
- Michael Hummel – Charité, Berlin
- Dirk Jäger – UHH, Heidelberg
- Jens Krüger – EKUT, Tübingen
- Nisar Malek – UKT, Tübingen
- Thomas Meitinger – HMGU & TUM, München
- Wolfgang E. Nagel – TU Dresden, Dresden
- Julio Saez-Rodriguez – UHH, Heidelberg
- Christoph Schickhardt – UHH, Heidelberg
- Thomas Keane – EMBL-EBI Cambridge, UK
- Mario Fritz & Ninja Marnau – CISPA Saarbrücken
- Stephan Hachinger – LRZ München
- Alice McHardy – HZI Braunschweig
- Stefan Fröhling & Hanno Glimm – National Center for Tumor Diseases (NCT), Heidelberg and Dresden